ДНК – молекула, которая в длину может быть равна росту взрослого человека. Чтобы её можно было поместить в клетку, существуют различные способы ее упаковки. Сначала она наматывается, как нитка на йо-йо, на нуклеосомы – структуры, состоящие из 8 специальных белков – гистонов. После в некоторых местах специфические белки связываются с ДНК, в результате формируя петли. Далее ДНК компактизуется еще сильнее, образуя хроматиды и хромосомы. Все эти структуры формируют пространственную организацию генома.
Изучение пространственной организации генома человека помогает лучше понимать, как именно регулируется работа генов. Для таких исследований используется метод Hi-C. Он позволяет получить картину контактов всех фрагментов генома друг с другом на всех структурных уровнях: от самого низкого (петель между регуляторными участками) до самого высокого (хромосомных территорий и целого ядра).
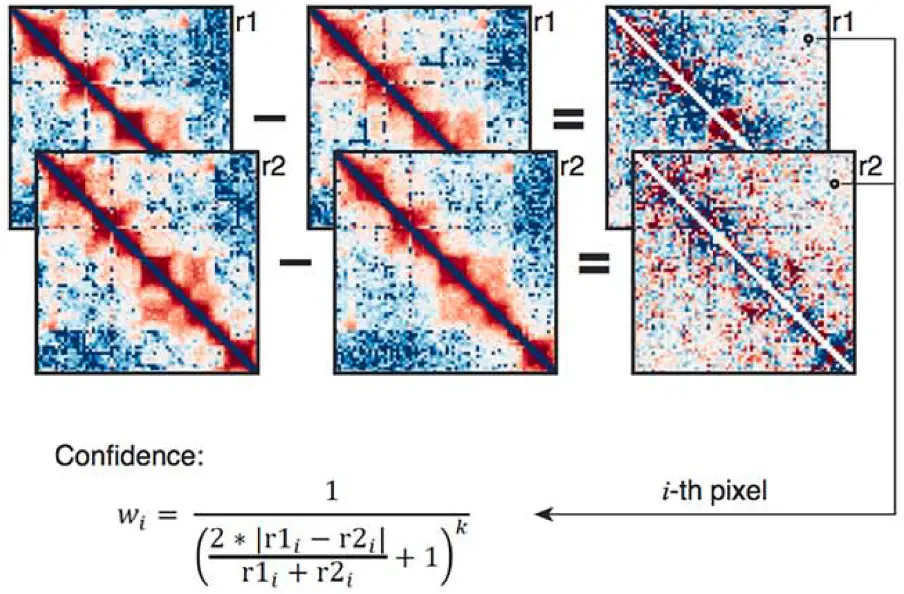
Хотя Hi-C считается довольно точным методом изучения трёхмерного генома, не лишён он и недостатков. Сейчас для проведения Hi-C существуют уже готовые наборы со всеми необходимыми реагентами и прописью процедур. Но провести исследование по стандартному протоколу не всегда представляется возможным: иногда материала бывает значительно меньше, чем требуется по стандарту, или – наоборот – требуется изучить слишком «объемный» объект, как, например, ткани мозга или образцы, полученные биопсией. В таких случаях исследователи дорабатывают протоколы с учётом особенностей материала и своих нужд, но полученные результаты становятся сложнее сравнивать с теми, что были получены по стандартным протоколам. Некоторые группы исследователей уже разработали подходы, которые учитывали бы такие погрешности, однако они все еще недостаточно точны.
Сотрудники биологического факультета МГУ в составе междисциплинарной команды исследователей из Сколтеха и НИЦ «Курчатовский институт» разработали новый метод нормализации данных Hi-C, который заметно снижает погрешности и их влияние на результаты – биоинформатический алгоритм HiConfidence. Особенность этого подхода заключается в том, что он учитывает силу и устойчивость взаимодействия внутри каждой отдельно взятой пары фрагментов генома. В результате данные, плохо воспроизводящиеся в повторах эксперимента, практически не влияют на финальные результаты, что позволяет значительно повысить качество получаемых карт пространственной организации генома и облегчить их интерпретацию.
Проверку качества работы HiConfidence проводили на данных, полученных при анализе пространственной организации генома клеток человека и плодовой мушки (Drosophila melanogaster). Применение инструмента HiConfidence позволило заметно снизить уровень «шума» в исходных данных. В результате удалось проанализировать влияние изменений ацетилирования (присоединения ацетиленовых групп) гистонов на плотность укладки геномной ДНК на масштабе отдельных геномных областей и хромосом в целом.
«Анализ больших данных в геномике и сравнение результатов, полученных в разных лабораториях, часто осложнены погрешностями, вносимыми при проведении экспериментов. Наличие биоинформатического инструмента, нивелирующего эти погрешности – вне зависимости от их конкретной природы – значительно облегчит выполнение проектов, в которых необходимо перекрёстное сравнение большого числа данных, полученных разными людьми в разное время»,
— подытожил один из авторов исследования, ведущий научный сотрудник кафедры молекулярной биологии биологического факультета МГУ Сергей Ульянов.


