Молекулярная биология простейших
Направления исследований
Группа занимается изучением структуры и функций генома жгутиковых простейших, относящихся к семейству Trypanosomatidae (класс Kinetoplastea).
Представители данного семейства являются облигатными паразитами, в процессе эволюции освоившими необычайно широкий круг хозяев: других простейших и высшие растения, членистоногих и позвоночных.
Используемые методы
- Стандартные методы молекулярной биологии
- Работа с культурами клеток простейших
- Фракционирование органелл
-
Методы современной биоинформатики
Работа с данными NGS, сравнительная геномика
- Программирование, разработка алгоритмов анализа данных
Руководитель группы
Сотрудники
Студенты 22/23
- Новожилова Татьяна Сергеевна, аспирант
- Конакова Зоя Александровна, студент-бакалавр
Сотрудничество с зарубежными лабораториями
Группа сотрудничает с лабораторий Геномики простейших под руководством Вячеслава Юрченко (Остравский государственный университет, Чехия).
Молекулярная биология простейших
Кинетопластиды получили своё название за наличие уникальной клеточной органеллы (кинетопласта), возникшей в результате эволюции митохондриального аппарата клетки. Рано отделившийся от общего древа эукариот филум накопил (или сохранил) ряд уникальных черт молекулярной организации, буквально меняющих общепринятые преставления о функционировании генома.
В фокусе внимания нашей научной группы находится механизм редактирования митохондриальных транскриптов.
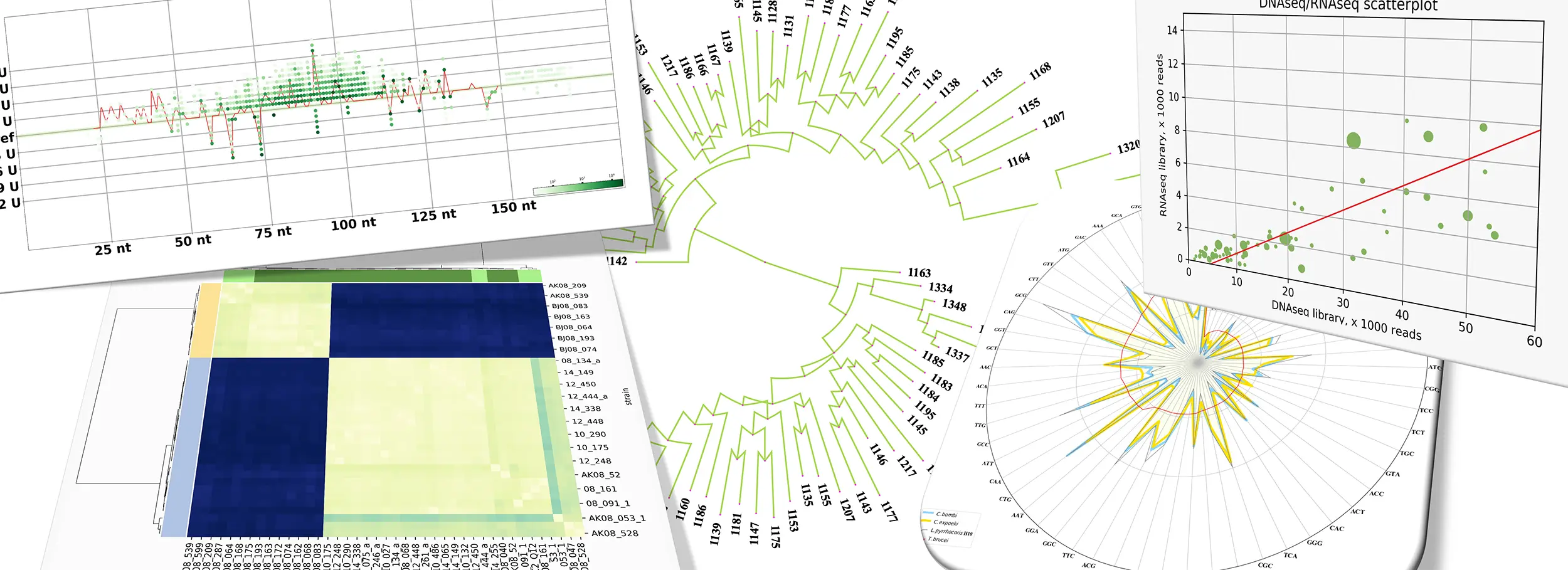
Это сложная генетическая система, позволяющая интегрировать информацию с разных локусов генома в одной матричной РНК, обеспечивая таким образом дополнительную вариабельность (аналогично альтернативному сплайсингу), и создавая дополнительный уровень контроля экспрессии генов. В процессе редактирования первичный транскрипт определенного участка митохондриальной ДНК (криптогена) претерпевает множественные вставки\удаления уридилов (U) мультиферментными комплексами при участии коротких молекул РНК, называемых гидовыми. Это короткие РНК определяют сайты вставки\удаления оснований, таким образом, привнося генетическую информацию с других локусов. Так как в редактировании одного криптогена обычно участвуют десятки разных гРНК, процесс может приводить к образованию целой серии различных продуктов.
Благодаря развитию технологий NGS, а также разработке нами специализированного программного обеспечения для анализа данных T-Aligner, нам удалось изучить разнообразие транскриптов в митохондриях кинетопластид, а также описать локальное альтернативное редактирование (процесс формирования изоформ мРНК за счет нестрого соответствия структуры гРНК и редактируемого ей региона мРНК, допускающего вариативность сайтов вставки U). Логично предположить, что разнообразие возможных транскриптов напрямую зависит от числа генов гРНК, и действительно, репертуар генов гРНК различается у разных видов.
Мы активно занимаемся изучением эволюции генов гРНК и роли системы редактирования с жизненным циклом клеток простейших в целом.
По-видимому, эволюция системы редактирования РНК привела к образованию кинетопласта как структуры и разделению митохондриальной ДНК на кольцевые популяции двух типов: максикольца и миникольца. В настоящее время (опять же, благодаря развитию технологий секвенирования) существенно расширился набор изученных видов. Стало понятно, что у более «современных» родов разнообразные по первичной структуре миникольца являются исключительными носителям гРНК, тогда как у более «древних» гРНК находится на относительно крупных молекулах.
Поскольку именно развитие системы миниколец могло обусловить формирование уникального дисковидного ассоциата ДНК (кинетопластной ДНК), мы также активно изучаем белковые факторы, играющие роль в его укладке и обеспечивающие транскрипцию. В геноме трипаносоматид обнаруживается семейство генов, кодирующих относительно небольшие белки с HMG доменами, направляющиеся в митохондрию. Эти белки получили название KAP (Kinetoplast Associated Proteins), так как многие из них выделялись в составе нуклеоида митохондрии. Мы устанавливаем роль индивидуальных KAP в обеспечении функционирования митохондриального генома простейших.
Геномика простейших
В последние годы в нашей группе активно развивается геномное направление.
Мы занимается полногеномным сравнительным анализом не только митохондриального, но и ядерного генома кинетопластид, а также геномов вирусов и эндосимбионтов, встречающихся в их клетках.
Методами современной биоинформатики мы пытаемся выявить «горячие» точки в геномах кинетопластид и изучить механизмы генетической изменчивости, обеспечившей им невероятный уровень адаптации к своим хозяевам.
Например, мы ведем поиска факторов вирулентности лейшманий (наиболее известных патогенов человека), сравнивая их с ближайшими родственниками, не способными паразитировать на теплокровных хозяевах. Как ни странно, одним из важных факторов, способствующих вирулентности клетки, является наличие в ней особого вируса лейшманий, взаимодействием которого с клеткой паразита мы с коллегами активно занимаемся.
Публикации сотрудников группы
- Complete minicircle genome of Leptomonas pyrrhocoris reveals sources of its non-canonical mitochondrial RNA editing events. Nucleic Acids Research, 2021 (http://dx.doi.org/10.1093/nar/gkab114)
- The Remarkable Metabolism of Vickermania ingenoplastis: Genomic Predictions. Pathogens, 2021 (http://dx.doi.org/10.3390/pathogens10010068)
- Common structural patterns in the maxicircle divergent region of trypanosomatidae. Pathogens, 2020 (http://dx.doi.org/10.3390/pathogens9020100)
- Genomic variation among strains of Сrithidia bombi and С. expoeki. mSphere, 2019 (http://dx.doi.org/10.1128/msphere.00482-19)
- Gene expression to mitochondrial metabolism: Variability among cultured Trypanosoma cruzi strains. PLoS ONE, 2018.
- Trypanosomatid mitochondrial RNA editing: dramatically complex transcript repertoires revealed with a dedicated mapping tool. Nucleic Acids Research, 2017 (http://dx.doi.org/10.1093/nar/gkx1202)
- Minicircle kinetoplast genome of insect trypanosomatid Leptomonas pyrrhocoris. Biochemistry, 2017.
- Genome of Leptomonas pyrrhocoris: a high-quality reference for monoxenous trypanosomatids and new insights into evolution of Leishmania. Scientific reports, 2016. (http://dx.doi.org/10.1038/srep23704)
- Gene loss and error-prone RNA editing in the mitochondrion of Perkinsela, an endosymbiotic kinetoplastid. mBio, 2015. (http://dx.doi.org/10.1128/mbio.01498-15)
- Diversity of mitochondrial genome organization. Biochemistry, 2012. (http://dx.doi.org/10.1134/S0006297912130020)
- From cryptogene to gene? ND8 editing domain reduction in insect trypanosomatids. European Journal of Protistology, 2012. (http://dx.doi.org/10.1016/j.ejop.2011.09.002)
- Selective amplification of maxicircle classes during the life cycle of Leishmania major. Molecular and Biochemical Parasitology, 2009. (http://dx.doi.org/10.1016/j.molbiopara.2009.01.014)
- The kinetoplast genome of Leishmania major contains several maxicircle classes undergoing differential amplification at the amastigote stage. Protistology, 2007.
- Mitochondrial RNA editing in Trypanoplasma borreli: New tools, new revelations / Computational and Structural Biotechnology Journal, 2022 (http://dx.doi.org/10.1016/j.csbj.2022.11.023)
- Trypanosoma cruzi strain and starvation-driven mitochondrial RNA editing and transcriptome variability. RNA, 2022. (http://dx.doi.org/10.1261/rna.079088.121)
- Analyses of Leishmania-LRV co-phylogenetic patterns and evolutionary variability of viral proteins. Viruses, 2021. (http://dx.doi.org/10.3390/v13112305)
- Catalase impairs Leishmania mexicana development and virulence. Virulence, 2021. (http://dx.doi.org/10.1080/21505594.2021.1896830)
- Ku80 is involved in telomere maintenance but dispensable for genomic stability in Leishmania mexicana. PLoS Neglected Tropical Diseases, 2021. (http://dx.doi.org/10.1371/journal.pntd.0010041)
- Genome analysis of Endotrypanum and Porcisia spp., closest phylogenetic relatives of Leishmania, highlights the role of amastins in shaping pathogenicity. Genes, 2021 (http://dx.doi.org/10.3390/genes12030444)